檢測到您當前使用浏覽器版本過于老舊,會導緻無法正常浏覽網站;請您使用電腦裡的其他浏覽器如:360、QQ、搜狗浏覽器的極速模式浏覽,或者使用谷歌、火狐等浏覽器。
 下載Firefox
下載Firefox
檢測到您當前使用浏覽器版本過于老舊,會導緻無法正常浏覽網站;請您使用電腦裡的其他浏覽器如:360、QQ、搜狗浏覽器的極速模式浏覽,或者使用谷歌、火狐等浏覽器。
 下載Firefox
下載Firefox
1988年,加州大學洛杉矶分校Michael Grunstein教授團隊發現,真核細胞中的核小體不僅僅是供DNA纏繞的結構,它在調節基因表達上還有着重要作用【1】。此後,随着表觀遺傳學先驅David Allis教授在1996年對組蛋白尾部修飾酶的突破性發現【2】和2001年提出的Histone Code假說【3】,人們對染色質結構與基因轉錄調控之間關系的理解進入了嶄新的一頁。在一系列組蛋白新修飾被鑒定【4】的同時,人們迫切地想知道這些特異性修飾的性質和功能有何異同,又是如何調控基因表達?例如,越來越多的證據表明,來自環境的代謝物會通過染色質的修飾實現基因轉錄調控【5】,但作為表觀遺傳調控的重要分子基礎,很多修飾對基因表達調控的影響尚不清楚,亟需對這些多層次的表觀遺傳信息加以解析和關聯。
2023年3月2日,beat365官方网站季雄組與化學與分子工程學院陳鵬組合作在Cell雜志發表了題為“Linking chromatin acylation mark-defined proteome and genome in living cells”的研究論文,發展了在活細胞内“關聯解析”蛋白質修飾機制與功能的“單位點-多組學”技術-SiTomics,揭示了受染色質酰化修飾介導的豐富的互作組學信息,建立了表觀遺傳調控的蛋白質組與基因組“信息關聯”。

傳統的生物學方法往往借助抗體識别原位研究蛋白質的翻譯後修飾,對抗體的特異性具有很高要求,為克服這些局限性,本研究發展了一種具有單氨基酸位點分辨率的多組學技術Single-site-resolved multi-omics(SiTomics,圖1)。通過将一系列帶有賴氨酸酰化修飾的“光交聯”非天然氨基酸,以位點特異的方式引入活細胞内的組蛋白當中,原位模拟内源修飾在基因組上的分布,并與蛋白質光交聯和組學鑒定技術、基因組測序技術等相結合,系統性地開展了由位點特異的修飾介導的染色質相互作用蛋白質組與基因組“關聯鑒定”。
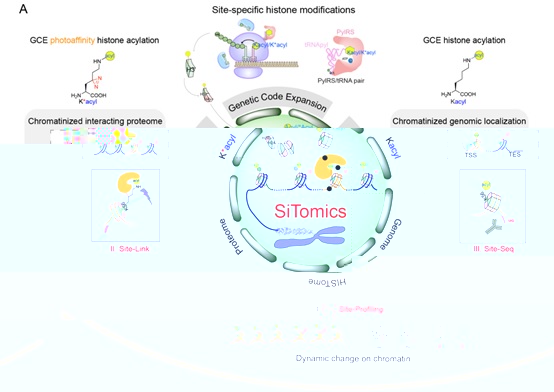
圖1“關聯解析”染色質動态修飾機制與功能的“單位點-多組學”技術-SiTomics (Single-site-resolved multi-omics)。
I. Site-Profiling: 單位點修飾的動态變化情況的比較, II. Site-Link: 通過遺傳密碼子拓展技術在組蛋白上引入K*acyl,利用光交聯蛋白質組學鑒定單位點修飾的相互作用蛋白質組, III. Site-Seq:通過遺傳密碼子拓展技術在組蛋白上引入Kacyl,結合傳統ChIP-seq技術研究具有修飾的核小體在基因組中所處的位置。
“單位點分辨”的動态修飾定量鑒定
組蛋白修飾位點衆多,不同位點對代謝物響應程度的定量比較,會因不同多肽對質譜響應的差異而難以實現。如組蛋白H3K56處在DNA纏繞進出口的位置,在複制、轉錄和修複等過程中具有重要意義,由于該位點抗體效果差,研究進展緩慢。在之前的研究中,人們往往利用同位素标記的代謝物處理細胞并鑒定同位素标記的修飾肽段,但這樣的方法會損失一些動态變化信息,尤其是對修飾量減少或者原有修飾經擦除後重新被修飾的情況,都難以提供豐度信息。利用SILAC技術标記賴氨酸和精氨酸,整合平行比較同樣長度、修飾和電荷數的同一肽段的變化情況,可以更為精準地定量比較修飾的動态變化(Site-Profiling, I)。最終,通過加權分析鑒定出H3K56位點在短鍊脂肪酸(如巴豆酸、b-羟基丁酸)處理下,都顯著地發生了相應的酰化修飾。
互作蛋白質組與染色質分布的“協同解析”
将Site-Link技術應用于H3蛋白的K37, K56和K79等位點(H3K*56ac,H3K*56cr,H3K*56bhb,H3K*56PABK*,H3K*37cr,H3K*79cr),利用對這些帶有光交聯探針的組蛋白進行交聯-富集和質譜鑒定,獲得了不同位點/修飾的相互作用蛋白質組信息,發現不同“位點-修飾”組合的确具有獨特的相互作用蛋白質組。
接下來,作者對巴豆酰化修飾進行了細緻的研究(圖2)。與H3K4me3一樣,賴氨酸的巴豆酰化修飾(Kcr)被報道分布于基因組啟動子區域。将SiTomics技術應用于H3K56cr,确實捕獲到一系列報道與H3K4me3相互作用的蛋白質,随機選取H3K4me3在基因組中分布的位點驗證H3K56cr也分布在這些位置。另外,研究發現H3K56cr具有一個非常特異的相互作用蛋白GLYR1,與H3K56位的其他修飾基本都沒有結合。鑒于之前的報道顯示GLYR1主要分布在基因内部(gene body)區域,作者推斷GLYR1可能與巴豆酰化在基因内部的相對高豐度分布有關,這也在一定程度上解釋了H3K56cr是先被整合進入基因組中,再與GLYR1産生相互作用,作者進一步推斷細胞中可能存在着“正交”的“識别-裝載”系統,能夠協助這些攜帶有翻譯後修飾的組蛋白整合進入染色質的相應位置,從而使這些組蛋白能夠“重現”内源修飾在基因組上的分布,其獲得的組學數據便是染色質化的蛋白質組和基因組學信息。為驗證這一論斷,作者随即對賴氨酸的β-羟基丁酰化修飾(Kbhb)進行了更為系統的研究。
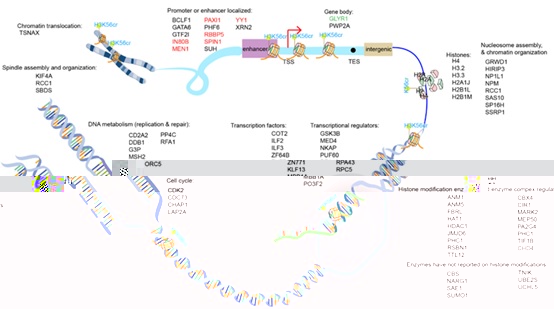
圖2 SiTomics技術“協同解析”受H3K56cr介導的“ 染色質化(Chromatinized)”相互作用蛋白質組與基因組。
β-羟基丁酰化是與“生酮飲食法”(Keto Diet )最為密切的一個化學修飾,但目前對其的研究報道甚少。将Site-Seq技術應用于β-羟基丁酰化,作者發現帶有H3K56bhb修飾的組蛋白并非随機地分布在染色質中,而無修飾的H3K56則相對随機分布。H3K56bhb确實可以增加一些染色質位點的開放性,進一步的Hi-C測序發現,單位點修飾的H3K56bhb便足以引起染色體三維結構的變化(圖3)。
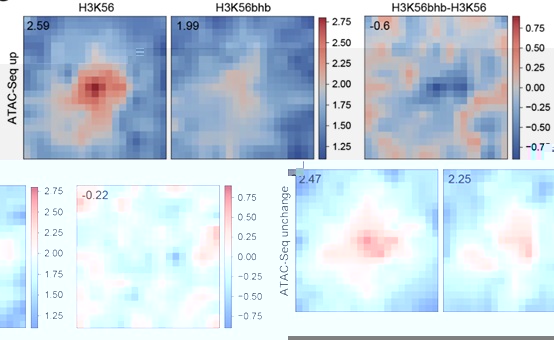
圖3 H3K56bhb單位點修飾對三維基因組的影響
“單位點-多組學”技術揭示生理情況的染色體調控
在确認了該技術對生理狀态的模拟之後,作者們接下來将其應用于酮體代謝下的染色質調控研究。首先通過免疫熒光發現酮體代謝下的β-羟基丁酰化修飾在細胞核中呈現特異的聚集性分布,這可能提示其在細胞核中獨特的調控作用。為解析其機制,酮體代謝情況下,H3K56bhb修飾會顯著增加,則可利用SiTomics工具的“單位點-多組學”優勢研究H3K56bhb的相互作用蛋白,并檢測在基因組上是否存在特異分布。結果發現H3K56bhb與BRD4存在相互作用,同時,H3K56bhb在超級增強子中有顯著分布(圖4),而作為對照的H3K9bhb則幾乎沒有分布。作者因此推測可能酮體代謝情況下的染色體調控有超級增強子的參與。
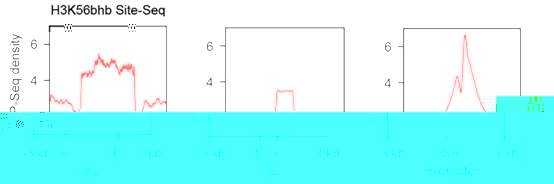
圖4 Site-Seq 分析H3K56bhb在基因組的特異分布
最後,通過利用Nabhb處理細胞、模拟酮體代謝下細胞内的染色質動态調控情況,結果發現超級增強子的确發生了顯著變化,且與H3K56bhb的基因組整合位點相一緻。此外,β-羟基丁酸處理下,受影響的超級增強子附近的基因其Pol II結合增加,而在H3K56bhb引入的細胞中,這些基因附近的Pol II結合也明顯增加。進一步的RNA-seq分析證實這些發生變化的超級增強子可以幫助解釋酮體代謝情況下相關基因的高表達現象,從而在分子水平上實現了“代謝-修飾-調控”軸的貫穿(圖5)。

圖5 SiTomics 解析代謝情況下H3K56bhb定點修飾對細胞超級增強子的影響
綜上所述,該工作使用SiTomics技術系統鑒定了多種組蛋白修飾在染色質的互作蛋白組,并結合多種組學技術系統鑒定了H3K56bhb對染色質結構和基因表達的影響,并為深入開展蛋白質動态修飾的機制研究提供了強大的工具平台。揭示了超級增強子(super-enhancer)在細胞“代謝-修飾-基因轉錄”調控軸中發揮的重要作用。
beat365官方网站季雄研究員、北京大學化學與分子工程學院陳鵬教授、北大-清華生命科學聯合中心畢業生秦芳菲博士是本文的共同通訊作者。秦芳菲博士、北京大學PTN項目畢業生李伯源博士為共同第一作者,beat365王輝博士等其它作者為本研究做出重要貢獻。該工作得到北大-清華生命科學聯合中心、科技部國家重點研發計劃、國家自然科學基金、啟東創新基金和細胞增殖與分化教育部重點實驗室等資助。感謝北京大學鳳凰工程多個儀器平台對本項目的大力支持。
季雄課題組長期從事RNA聚合酶亞基功能調控和疾病機理的研究。主要集中在RNA聚合酶相關的非經典功能調控、疾病和分子探針等方向,近期成果發表在Cell、Molecular Cell (2)、Genome Biology (2)、Nature Communications、Cell Discovery、CMLS和iScience等雜志上,為選擇性基因表達調控提供新的假說。現因發展需要,招聘博士後2-3名。http:/homes/Index/news_cont_jl/16/545.html
原文鍊接:https://www.cell.com/cell/fulltext/S0092-8674(23)00109-5
參考文獻:
1. Han, M., and Grunstein, M. (1988). Nucleosome loss activates yeast downstream promoters in vivo. Cell 55, 1137-1145.
2. Brownell, J.E., Zhou, J., Ranalli, T., Kobayashi, R., Edmondson, D.G., Roth, S.Y., and Allis, C.D. (1996). Tetrahymena histone acetyltransferase A: a homolog to yeast gcn5p linking histone acetylation to gene activation. Cell 84, 843-851.
3. Jenuwein, T., and Allis, C.D. (2001). Translating the histone code. Science 293, 1074-1080.
4. Huang, H., Sabari, B.R., Garcia, B.A., Allis, C.D., and Zhao, Y. (2014). SnapShot: histone modifications. Cell 159, 458-458.e451.
5. Sabari, B.R., Zhang, D., Allis, C.D., and Zhao, Y. (2017). Metabolic regulation of gene expression through histone acylations. Nat Rev Mol Cell Biol 18, 90-101.