檢測到您當前使用浏覽器版本過于老舊,會導緻無法正常浏覽網站;請您使用電腦裡的其他浏覽器如:360、QQ、搜狗浏覽器的極速模式浏覽,或者使用谷歌、火狐等浏覽器。
 下載Firefox
下載Firefox
檢測到您當前使用浏覽器版本過于老舊,會導緻無法正常浏覽網站;請您使用電腦裡的其他浏覽器如:360、QQ、搜狗浏覽器的極速模式浏覽,或者使用谷歌、火狐等浏覽器。
 下載Firefox
下載Firefox
2023年4月13日,beat365官方网站陸劍課題組在Molecular Biology and Evolution在線發表題為Adaptive evolution of the Spike protein in coronaviruses的論文,發現新冠病毒Spike(S)基因在持續演化以及冠狀病毒長期演化過程中均受到強烈的正選擇,但是正選擇靶點則存在顯著差異,揭示了冠狀病毒S基因的演化規律。
冠狀病毒是一種正義單鍊的RNA病毒,在自然界中廣泛存在,分為Alpha,Beta,Gamma和Delta四個屬,可以感染哺乳動物和鳥類等多個物種。目前已知有七種冠狀病毒可以感染人類,包括當前正在流行的新冠病毒。冠狀病毒的Spike(S)蛋白通過與宿主細胞表面的受體相互作用,促進病毒侵染細胞。S蛋白可以分為S1和S2區域,S1識别并與宿主細胞表面的受體結合,S2促進病毒與宿主細胞的膜融合。S1在決定宿主範圍和組織嗜性方面起着至關重要的作用,可以進一步分為N端結構域(S1-NTD)和C端結構域(S1-CTD),兩者都可以作為受體結合結構域(Receptor binding domain,RBD)與宿主受體結合。S蛋白還是宿主中和抗體的關鍵靶點。因此,病毒和宿主之間的演化軍備競賽(evolutionary arms race)将驅動S蛋白序列快速演化。
大多數冠狀病毒的宿主為動物(圖1a)。本研究對四個屬的冠狀病毒基因組序列進行了分子演化分析,發現S1-NTD在四個屬的冠狀病毒演化過程中存在明顯的正選擇信号。S1-NTD主要識别宿主細胞的糖受體,也可以識别宿主細胞的輔助蛋白,因此S1-NTD的正選擇信号可能來自于受體使用的多樣化,反映出冠狀病毒在不斷探測以适應不同宿主物種和組織的能力。此外,S1-NTD也可作為冠狀病毒宿主生物抗體的中和表位,冠狀病毒可能迅速演化以逃避宿主的免疫反應(圖1a)。
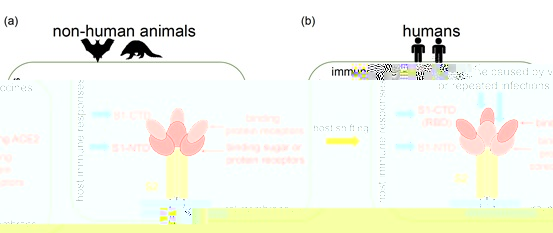
圖1. SARS-CoV-2和其他冠狀病毒之間S基因選擇壓力差異的模型 (Tang et al. 2023, MBE)
與冠狀病毒S基因的長期演化趨勢不同,本研究發現在新冠病毒的持續演化過程中,S基因的S1-CTD(RBD)受到了強烈的正選擇。通過對各突變株基因組序列的比較,正選擇信号主要集中在SARS-CoV-2的S1-CTD(RBD)區域,然而新冠病毒的S1-NTD區域并沒有檢測到正選擇信号。研究推測可能是宿主環境的變化以及人類免疫反應的選擇壓力導緻了新冠病毒與其他冠狀病毒正選擇靶點的變化(圖1b)。此研究也印證了陸劍團隊先前(2022年3月2号)與吳仲義|呂雪梅團隊在Molecular Biology and Evolution發表的文章“The runaway evolution of SARS-CoV-2 leading to the highly evolved Delta strain”的觀點,該研究認為随着新冠病毒感染人數的增加,病毒群體大小增加,提高适應性變異出現和固定的概率,加速适應性演化速率加快病毒的擴散,從而形成群體大小和演化速率之間的正反饋循環,導緻高度演化變異株的快速湧現(圖2)。這兩項研究均表明感染人數的急劇增大是促進病毒變異加快的重要因素。
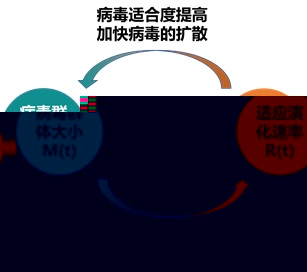
圖2 新冠病毒感染人數與演化速率之間的正反饋 (Ruan et al. 2022, MBE)
綜上所述,本研究發現S基因,特别是S1區域,在SARS-CoV-2和其他冠狀病毒中都經曆了強烈的正選擇。雖然S1-NTD在所有四個冠狀病毒屬中都表現出正選擇,但在SARS-CoV-2的持續演化中,主要在S1-CTD(RBD)中檢測到正選擇信号,這可能是由于宿主環境的改變以及人類群體大規模感染所産生的免疫壓力所緻。本研究有助于加深對病毒演化規律的理解,也為疫苗靶點的設計提供了參考。
陸劍教授為該研究的通訊作者,生科院博士後唐小鹿為第一作者,醫科院病原所錢朝晖研究員和中科院昆明動物所呂雪梅研究員也為該工作做出了重要貢獻。該工作得到了國家科技部、北京市自然科學基金委和beat365官方网站啟東産業創新基金等的支持。
原文鍊接: https://doi.org/10.1093/molbev/msad089
原文鍊接: https://doi.org/10.1093/molbev/msac046